主成分分析(PCA)は通常、共分散行列の固有分解によって説明されます。ただし、データ行列特異値分解(SVD)を介して実行することもできます。どのように機能しますか?これら2つのアプローチの関係は何ですか?SVDとPCAの関係は何ですか?
または、言い換えると、データ行列のSVDを使用して次元削減を実行する方法ですか?
主成分分析(PCA)は通常、共分散行列の固有分解によって説明されます。ただし、データ行列特異値分解(SVD)を介して実行することもできます。どのように機能しますか?これら2つのアプローチの関係は何ですか?SVDとPCAの関係は何ですか?
または、言い換えると、データ行列のSVDを使用して次元削減を実行する方法ですか?
回答:
データ行列サイズをます。ここで、はサンプル数、は変数の数です。それが中央にあると仮定しましょう。つまり、列の平均が差し引かれ、ゼロになりました。
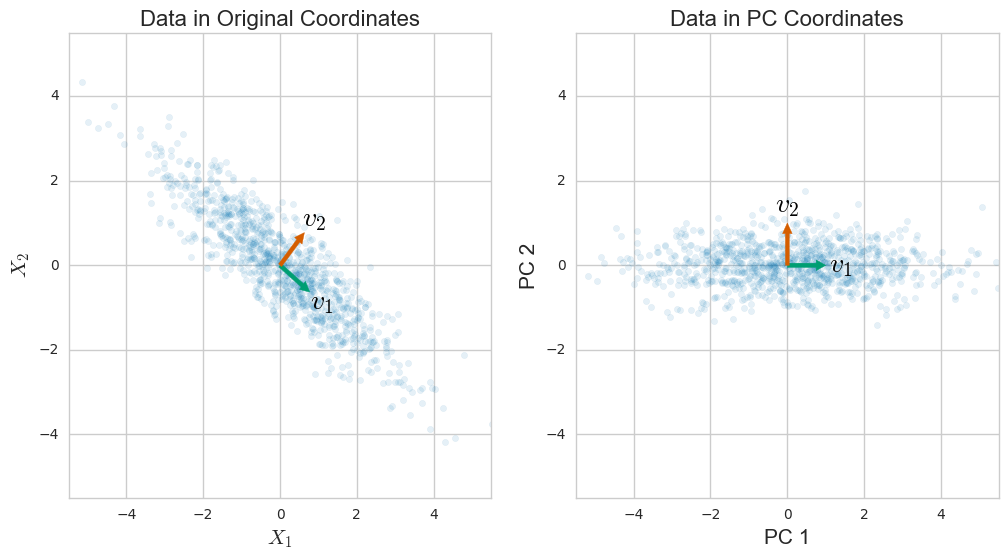
次に、共分散行列は、与えられます。これは対称行列であるため、対角化することができます:ここでは固有ベクトルの行列(各列は固有ベクトルです)およびは対角線上の降順の固有値をもつ対角行列。固有ベクトルは、データの主軸または主方向と呼ばれます。主軸上のデータの射影は主成分と呼ばれ、PCスコアとも呼ばれます
特異値分解を実行すると、分解ここで、はユニタリ行列で、は対角行列です特異値は。ここから、つまり、右特異ベクトルは主方向であり、特異値は介して共分散行列の固有値に関連することを意味します。。主成分は
要約する:
SVDとPCAとの直感的な関係は何である math.SE.で非常に人気があり、非常によく似たスレッドが-
データのSVDによるデータのPCAが必要な理由 -SVDを介してPCAを実行することの利点についての説明[短い答え:数値安定性]。
Biplotとの関係におけるPCAおよびコレスポンデンス分析 -一部の同種の手法のコンテキストにおけるPCA、すべてSVDに基づいています。
SVAがPCAより優れている点はありますか?-PCAの代わりにSVDを使用する利点があるかどうかを尋ねる質問[短い答え:不適切な質問]。
主成分分析、固有ベクトル、固有値を理解する-PCAの非技術的な説明を与える私の答え。注意を引くために、ここで1つの図を再現します。
@amoebaの回答に付随するPython&Numpyスニペットを作成しました。誰かに役立つ場合に備えて、ここに残しておきます。コメントは主に@amoebaの回答から取られています。
import numpy as np
from numpy import linalg as la
np.random.seed(42)
def flip_signs(A, B):
"""
utility function for resolving the sign ambiguity in SVD
http://stats.stackexchange.com/q/34396/115202
"""
signs = np.sign(A) * np.sign(B)
return A, B * signs
# Let the data matrix X be of n x p size,
# where n is the number of samples and p is the number of variables
n, p = 5, 3
X = np.random.rand(n, p)
# Let us assume that it is centered
X -= np.mean(X, axis=0)
# the p x p covariance matrix
C = np.cov(X, rowvar=False)
print "C = \n", C
# C is a symmetric matrix and so it can be diagonalized:
l, principal_axes = la.eig(C)
# sort results wrt. eigenvalues
idx = l.argsort()[::-1]
l, principal_axes = l[idx], principal_axes[:, idx]
# the eigenvalues in decreasing order
print "l = \n", l
# a matrix of eigenvectors (each column is an eigenvector)
print "V = \n", principal_axes
# projections of X on the principal axes are called principal components
principal_components = X.dot(principal_axes)
print "Y = \n", principal_components
# we now perform singular value decomposition of X
# "economy size" (or "thin") SVD
U, s, Vt = la.svd(X, full_matrices=False)
V = Vt.T
S = np.diag(s)
# 1) then columns of V are principal directions/axes.
assert np.allclose(*flip_signs(V, principal_axes))
# 2) columns of US are principal components
assert np.allclose(*flip_signs(U.dot(S), principal_components))
# 3) singular values are related to the eigenvalues of covariance matrix
assert np.allclose((s ** 2) / (n - 1), l)
# 8) dimensionality reduction
k = 2
PC_k = principal_components[:, 0:k]
US_k = U[:, 0:k].dot(S[0:k, 0:k])
assert np.allclose(*flip_signs(PC_k, US_k))
# 10) we used "economy size" (or "thin") SVD
assert U.shape == (n, p)
assert S.shape == (p, p)
assert V.shape == (p, p)
PCAから始めましょう。それぞれd個の数値(または次元)で構成されるn個のデータポイントがあるとします。このデータを中央に配置する(各データベクトルから平均データポイント引く)と、データを積み重ねて行列を作成できます。
共分散行列
データが与えられるさまざまな座標がどの程度変化するかを測定します。したがって、データの変動をキャプチャするために設計されたPCAが共分散行列の観点から与えられることは驚くことではないでしょう。特に、の固有値分解は次のようになります。
ここで、は番目の主成分、またはPCであり、は番目の固有値であり、番目のPCに沿ったデータの分散にも等しくなります。この分解は、線形代数の一般的な定理に由来し、リラティーノをPCAに動機付けるために、いくつかの作業を行う必要があります。
SVDは、列スペースと行スペースの観点からマトリックスを理解する一般的な方法です。(これは、行列を行と列の空間に直観的に関係する他の行列に関して書き換える方法です。)たとえば、行列ドメインと範囲で方向と見つけることができるので、
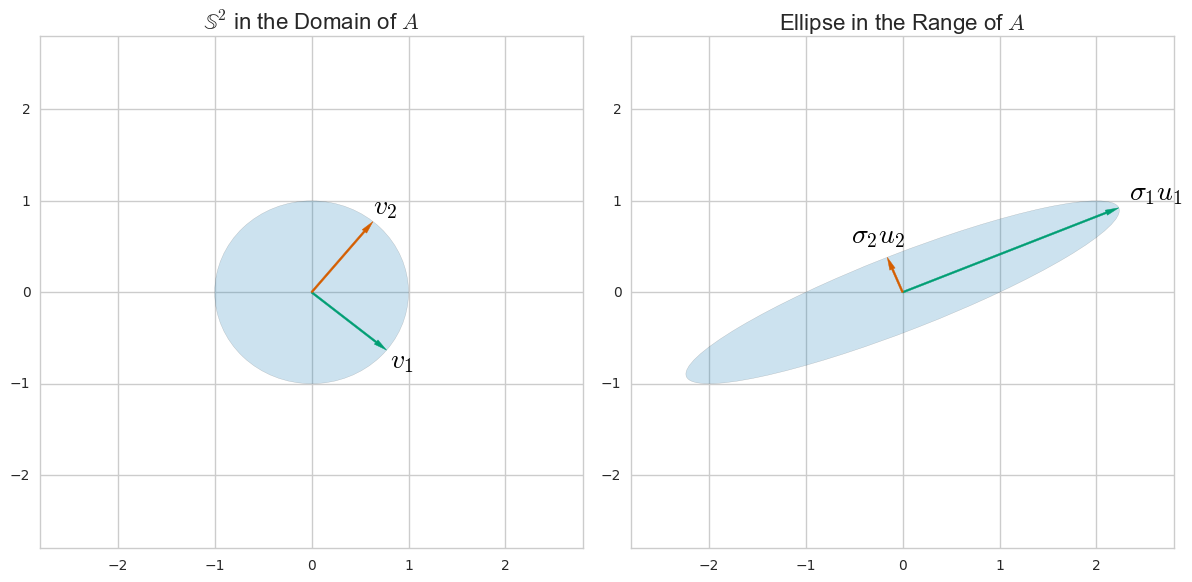
これらを見つけるには、線形変換としてのがそのドメイン内の単位球を楕円にモーフィングする方法を検討することにより、楕円の主要な半軸がおよびと整列する前画像です。
いずれにせよ、上記のデータ行列(実際には設定だけ)に対して、SVDは
ここでとの固有値分解とvectors.A比較の正規直交セットである「右特異ベクトル」ことを明らかにする PCに等しく、「右特異ベクトルは、」あります
そして、「特異値」は、次を介してデータ行列に関連しています
一般に、正しい特異ベクトルは列空間にまたがっています。この特定の場合、は、番目の主成分の方向へのデータスケーリングされた投影を与えます。一般に、左の特異ベクトルはの行空間に広がります。これにより、PCのようにデータに広がる正規直交ベクトルのセットが得られます。
この長い記事では、PCAとSVDの関係の詳細と利点について説明します。